Imitare le condizioni fisiologiche aiuta i ricercatori a trovare leganti metallici
I ricercatori hanno sviluppato un metodo per identificare piccole molecole che legano gli ioni metallici. Gli ioni metallici sono essenziali in biologia. Ma identificare con quali molecole – e soprattutto con quali piccole molecole – gli ioni metallici interagiscono può essere difficile.
Per separare i metaboliti per l'analisi, i metodi metabolomici convenzionali utilizzano solventi organici e pH bassi, che possono causare la dissociazione dei complessi metallici. Pieter C. Dorrestein dell'Università della California a San Diego e colleghi volevano tenere insieme i complessi per l'analisi imitando le condizioni native riscontrate nelle cellule. Ma se avessero utilizzato condizioni fisiologiche durante la separazione delle molecole, avrebbero dovuto riottimizzare le condizioni di separazione per ciascuna condizione fisiologica che volevano testare.
I ricercatori hanno invece sviluppato un approccio in due fasi che introduce condizioni fisiologiche tra una separazione cromatografica convenzionale e un’analisi spettrometrica di massa (Nat. Chem. 2021, DOI: 10.1038/s41557-021-00803-1). Innanzitutto, hanno separato un estratto biologico utilizzando la cromatografia liquida ad alte prestazioni convenzionale. Quindi hanno regolato il pH del flusso in uscita dalla colonna cromatografica per imitare le condizioni fisiologiche, hanno aggiunto ioni metallici e hanno analizzato la miscela con la spettrometria di massa. Hanno eseguito l'analisi due volte per ottenere spettri di massa di piccole molecole con e senza metalli. Per identificare quali molecole legano i metalli, hanno utilizzato un metodo computazionale che utilizza le forme dei picchi per dedurre le connessioni tra gli spettri delle versioni legate e non legate.
Un modo per imitare ulteriormente le condizioni fisiologiche, dice Dorrestein, sarebbe quello di aggiungere alte concentrazioni di ioni come sodio o potassio e basse concentrazioni del metallo di interesse. “Diventa un esperimento di competizione. Fondamentalmente ti dirà, OK, questa molecola in quelle condizioni ha più propensione a legare sodio e potassio o questo metallo unico che hai aggiunto", afferma Dorrestein. "Possiamo infondere molti metalli diversi contemporaneamente e possiamo davvero comprendere la preferenza e la selettività in quel contesto."
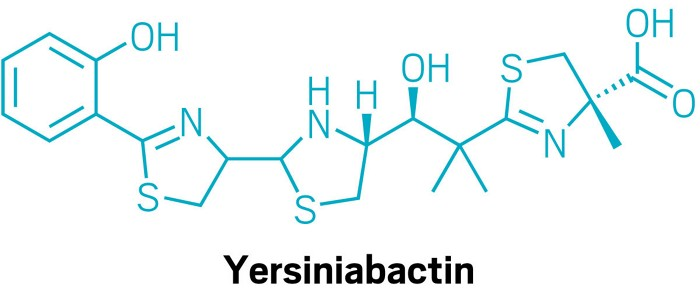
Negli estratti di coltura di Escherichia coli, i ricercatori hanno identificato composti noti che legano il ferro, come la yersiniabactina e l'aerobactina. Nel caso della yersiniabactina, hanno scoperto che può anche legarsi allo zinco.
I ricercatori hanno identificato composti che legano i metalli in campioni complessi quanto la materia organica disciolta nell’oceano. "È assolutamente uno dei campioni più complessi che abbia mai esaminato", afferma Dorrestein. “Probabilmente è complesso quanto, se non più complesso, del petrolio greggio”. Il metodo ha identificato l'acido domoico come una molecola che lega il rame e ha suggerito che si leghi al Cu2+ come un dimero.
"Un approccio omico per identificare tutti i metaboliti che legano i metalli in un campione è estremamente utile data l'importanza della chelazione biologica dei metalli", scrive in un articolo Oliver Baars, che studia i metaboliti che legano i metalli prodotti da piante e microbi alla North Carolina State University. e-mail.
"Dorrestein e colleghi forniscono un test elegante, molto necessario, per sondare meglio quale potrebbe essere il ruolo fisiologico degli ioni metallici nella cellula", scrive in una e-mail Albert JR Heck, un pioniere nelle analisi di spettrometria di massa nativa presso l'Università di Utrecht. “Un possibile passo successivo sarebbe quello di estrarre i metaboliti in condizioni native dalla cellula e frazionarli anche in condizioni native, per vedere quali metaboliti trasportano quali ioni metallici cellulari endogeni”.
Notizie di chimica e ingegneria
ISSN0009-2347
Copyright © 2021 Società Chimica Americana
Orario di pubblicazione: 23 dicembre 2021

